比对算法:修订间差异
小无编辑摘要 |
小无编辑摘要 |
||
| (未显示6个用户的7个中间版本) | |||
| 第7行: | 第7行: | ||
* 同源性只能用有没有来形容,不能说“70%同源”这种胡话,类似于“这个布尔函数<ref>布尔函数是信息技术课上的一种数据存储形式 | * 同源性只能用有没有来形容,不能说“70%同源”这种胡话,类似于“这个布尔函数<ref>布尔函数是信息技术课上的一种数据存储形式 | ||
只有0和正数两种值</ref>有80% | 只有0和正数两种值</ref>有80%是0”。 | ||
* | * 也就是说,一致度要求完全一样,相似度只要性质差不多也能算进去。 | ||
* | * 同源性是定性概念,一致度和相似度是定量概念。 | ||
== 比对类型 == | == 比对类型 == | ||
局部比对找出两个序列中高相似度的片段。 | |||
全局比对构建打分矩阵,打分…… | |||
半局部比对对一条序列使用局部比对,另一条使用全局比对,如此求得最大匹配度。 | |||
== 比对矩阵 == | == 比对矩阵 == | ||
对蛋白质或核酸序列进行比对,需要借助于矩阵(对于计分表的更高端叫法)打分,得分越高则代表二者越相似。 | |||
=== 核酸矩阵 === | === 核酸矩阵 === | ||
| 第63行: | 第63行: | ||
和上一张差不多 | 和上一张差不多 | ||
转换/颠换矩阵<ref>转换指的是嘌呤换嘌呤,嘧啶换嘧啶 | '''转换/颠换矩阵'''<ref>转换指的是嘌呤换嘌呤,嘧啶换嘧啶 | ||
颠换指的是嘌呤换嘧啶(这么基础感觉没必要多嘴啊w)</ref> | 颠换指的是嘌呤换嘧啶(这么基础感觉没必要多嘴啊w)</ref> | ||
| 第106行: | 第106行: | ||
PAM矩阵是目前蛋白质序列比较中最广泛使用的记分方法之一,基础的PAM-1 矩阵反应的是进化产生的'''每一百个氨基酸平均发生一个突变的量值(统计方法得到)'''。 | PAM矩阵是目前蛋白质序列比较中最广泛使用的记分方法之一,基础的PAM-1 矩阵反应的是进化产生的'''每一百个氨基酸平均发生一个突变的量值(统计方法得到)'''。 | ||
PAM-1是由相似度大于85% | PAM-1是由相似度大于85%的序列计算产生。 | ||
PAM-1 自乘n次,可以得到PAM- | PAM-1 自乘n次,可以得到PAM-n,即发生了多次突变。也就是说PAM越大,相似度越小。n即允许发生的点突变数。 | ||
==== BLOSUM矩阵 ==== | ==== BLOSUM矩阵 ==== | ||
BLOSUM 矩阵都是通过对大量符合特定要求的序列计算而来。如BLOSUM62表示计算了相似度>62% | BLOSUM 矩阵都是通过对大量符合特定要求的序列计算而来。如BLOSUM62表示计算了相似度>62%的蛋白质序列得到结果。 | ||
PAM基于全局比对,BLOSUM基于局部比对。 | |||
[[文件:屏幕截图(8).png|缩略图|比较PAM和BLOSUM]] | [[文件:屏幕截图(8).png|缩略图|比较PAM和BLOSUM]] | ||
==== GCM矩阵/遗传密码子矩阵 ==== | ==== GCM矩阵/遗传密码子矩阵 ==== | ||
两个氨基酸之间密码子所需的突变次数越多,替换代价越大。(多用于进化树) | |||
==== 疏水矩阵 ==== | ==== 疏水矩阵 ==== | ||
疏水性相近的氨基酸得分更高。 | |||
== 比对序列 == | == 比对序列 == | ||
=== 动态规划 === | === 动态规划 === | ||
核心原理是,如果你每一步都是最优解,那最后也一定是最优解。 | |||
也就是说,<u>因为你在列表走格子的时候每次都取了最优解,所以比对结果也应该是最优解</u>。 | |||
(有人知道这个点是怎么来的吗?道理上来说贪心算法不一定是全局最优) | |||
1950由理查德贝尔曼开发。 | |||
'''切记局部最优不是全局最优——<s>信息巨佬补</s>''' | |||
局部最优是贪心,全局才是动态规划 | |||
=== 打点法 === | === 打点法 === | ||
[[文件:序列比对笑传之compare cat brothers.png|缩略图|compare cat brothers]] | [[文件:序列比对笑传之compare cat brothers.png|缩略图|compare cat brothers]]将两条序列用这一张图中的方法分别列出,形成一个二维表格,在字母相同(实际上是氨基酸相同)的位置打上点。 | ||
如果打点后产生了对角线或者与其相同的平行线,这些区域具有一致性。 | |||
可用于寻找序列中的重复片段。 | |||
许多名字里带有Dot的软件可以用打点法比对序列如Dnadot,(dotlet似乎属于SIB | 许多名字里带有Dot的软件可以用打点法比对序列如Dnadot,(dotlet似乎属于SIB | ||
=== 全局比对之Needleman-Swunch算法 === | === 全局比对之Needleman-Swunch算法 === | ||
1970年由名字里的二人开发。 | |||
换一种方法理解打点法,您会发现她很像一种打分,只不过是0/1 | 换一种方法理解打点法,您会发现她很像一种打分,只不过是0/1 | ||
[[文件:NStrue.png|缩略图|Needlemanswunch矩阵初始化]] | [[文件:NStrue.png|缩略图|Needlemanswunch矩阵初始化]] | ||
下面我们使用更加酷炫的动态规划(走格子),Needleman-Swunch的走格子步骤如下 | 下面我们使用更加酷炫的动态规划(走格子),Needleman-Swunch的走格子步骤如下 | ||
选取错配、匹配、空位对应的奖惩制度(注:上一个人编辑时用的错配和空位-1分,匹配+1分;此我为了讲述方便,后改为错配-1分、匹配+1分、空位-2分) | |||
1,初始化矩阵,将两条序列用上一张图中的方法分别列出并且在开头处空一格,形成一个二维表格,在表格的左上角的边缘打上0,-1,-2,。。。一直到表格结束 | 1,初始化矩阵,将两条序列用上一张图中的方法分别列出并且在开头处空一格,形成一个二维表格,在表格的左上角的边缘打上0,-1,-2,。。。一直到表格结束 | ||
2,方法:如图 | |||
<br>[[文件:比对算法举例.jpg|替代=错配-1分、匹配+1分、空位-2分|500x500像素|错配-1分、匹配+1分、空位-2分]] | |||
{{学科分类}} | |||
[[Category:生物信息学]] | |||
2026年1月9日 (五) 17:28的最新版本
介绍三种关于序列比对的算法,矩阵,进化树与更多
相似与一致与同源
- 一致度:如果两个序列(蛋白质或核酸)长度相同,那么它们的一致度定义为对应位置上相同的残基(氨基酸或碱基)的数目占总长度的百分比。
- 相似度:如果两个序列长度相同,则相似度定义为它们对应位置上相似的残基与相同的残基的数目和占总长度的百分数。
- 同源性只能用有没有来形容,不能说“70%同源”这种胡话,类似于“这个布尔函数[1]有80%是0”。
- 也就是说,一致度要求完全一样,相似度只要性质差不多也能算进去。
- 同源性是定性概念,一致度和相似度是定量概念。
比对类型
局部比对找出两个序列中高相似度的片段。
全局比对构建打分矩阵,打分……
半局部比对对一条序列使用局部比对,另一条使用全局比对,如此求得最大匹配度。
比对矩阵
对蛋白质或核酸序列进行比对,需要借助于矩阵(对于计分表的更高端叫法)打分,得分越高则代表二者越相似。
核酸矩阵
等价矩阵
将一致的计算为一分,不一致的计算为0分
| A | T | C | G | |
|---|---|---|---|---|
| A | 1 | 0 | 0 | 0 |
| T | 0 | 1 | 0 | 0 |
| C | 0 | 0 | 1 | 0 |
| G | 0 | 0 | 0 | 1 |
BLAST矩阵
将一致的计算为+4分,不一致的计算为-5分
和上一张差不多
转换/颠换矩阵[2]
将一致的计+1,转换计算-1,颠换计算-5
| A | T | C | G | |
|---|---|---|---|---|
| A | 1 | -5 | -5 | -1 |
| T | -5 | 1 | -1 | -5 |
| C | -5 | -1 | 1 | -5 |
| G | -1 | -5 | -5 | 1 |
蛋白质矩阵
PAM矩阵
PAM矩阵是目前蛋白质序列比较中最广泛使用的记分方法之一,基础的PAM-1 矩阵反应的是进化产生的每一百个氨基酸平均发生一个突变的量值(统计方法得到)。
PAM-1是由相似度大于85%的序列计算产生。
PAM-1 自乘n次,可以得到PAM-n,即发生了多次突变。也就是说PAM越大,相似度越小。n即允许发生的点突变数。
BLOSUM矩阵
BLOSUM 矩阵都是通过对大量符合特定要求的序列计算而来。如BLOSUM62表示计算了相似度>62%的蛋白质序列得到结果。
PAM基于全局比对,BLOSUM基于局部比对。

GCM矩阵/遗传密码子矩阵
两个氨基酸之间密码子所需的突变次数越多,替换代价越大。(多用于进化树)
疏水矩阵
疏水性相近的氨基酸得分更高。
比对序列
动态规划
核心原理是,如果你每一步都是最优解,那最后也一定是最优解。
也就是说,因为你在列表走格子的时候每次都取了最优解,所以比对结果也应该是最优解。
(有人知道这个点是怎么来的吗?道理上来说贪心算法不一定是全局最优)
1950由理查德贝尔曼开发。
切记局部最优不是全局最优——信息巨佬补
局部最优是贪心,全局才是动态规划
打点法
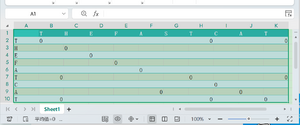
将两条序列用这一张图中的方法分别列出,形成一个二维表格,在字母相同(实际上是氨基酸相同)的位置打上点。
如果打点后产生了对角线或者与其相同的平行线,这些区域具有一致性。
可用于寻找序列中的重复片段。
许多名字里带有Dot的软件可以用打点法比对序列如Dnadot,(dotlet似乎属于SIB
全局比对之Needleman-Swunch算法
1970年由名字里的二人开发。
换一种方法理解打点法,您会发现她很像一种打分,只不过是0/1
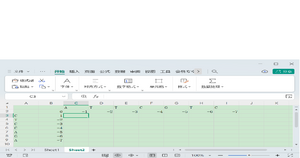
下面我们使用更加酷炫的动态规划(走格子),Needleman-Swunch的走格子步骤如下
选取错配、匹配、空位对应的奖惩制度(注:上一个人编辑时用的错配和空位-1分,匹配+1分;此我为了讲述方便,后改为错配-1分、匹配+1分、空位-2分)
1,初始化矩阵,将两条序列用上一张图中的方法分别列出并且在开头处空一格,形成一个二维表格,在表格的左上角的边缘打上0,-1,-2,。。。一直到表格结束

