染色质开放性检测技术:修订间差异
无编辑摘要 |
无编辑摘要 |
||
| 第44行: | 第44行: | ||
=== ATAC-Seq === | === ATAC-Seq === | ||
'''原理:'''通过使用高度活跃的突变体Tn5转座酶探测开放染色质,该突变体Tn5转座酶将测序接头插入基因组的开放区域。虽然天然存在的转座酶活性较低,但ATAC-seq使用突变的高活性转座酶。在一个称为“标签化”的过程中,Tn5转座酶使用测序接头切割并标记双链 DNA,然后对标记的DNA片段进行纯化、PCR扩增,并使用下一代测序进行测序。<br> | '''原理:'''通过使用高度活跃的突变体Tn5转座酶探测开放染色质,该突变体Tn5转座酶将测序接头插入基因组的开放区域。虽然天然存在的转座酶活性较低,但ATAC-seq使用突变的高活性转座酶。在一个称为“标签化”的过程中,Tn5转座酶使用测序接头切割并标记双链 DNA,然后对标记的DNA片段进行纯化、PCR扩增,并使用下一代测序进行测序。<br> | ||
'''特点:'''①快速高效:不需要像FAIRE-seq那样进行超声处理或苯酚-氯仿提取,没有像MNase-seq或DNase- | '''特点:'''①快速高效:不需要像FAIRE-seq那样进行超声处理或苯酚-氯仿提取,没有像MNase-seq或DNase-seq这样敏感的酶消化,整个工作可在三小时内完成;<br> | ||
②分辨率高:相比于其他三种方法,其操作位点更为精细,因此分辨率可以达到单核苷酸水平; | |||
abcdefg | abcdefg | ||
2024年8月5日 (一) 20:59的版本
前言
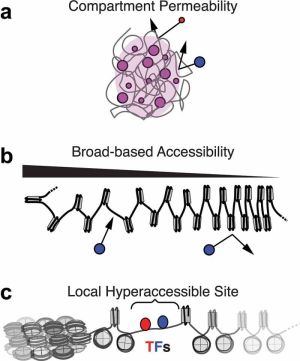
染色质开放性检测技术,又可称为染色质可及性检测技术,是用于观测蛋白质因子获得和维持对DNA的物理访问的控制能力(通常称为染色质可及性)的一类技术。对于“染色质可及性”这一术语,通常的基础教材停留于“常染色质”和“异染色质”这两种区分方法上,即可及性强为前者,反之为后者,这种划分方法简便且易于验证,常用的显带技术就可以做到这一点。然而,随着研究尺度不断深入,简单的二分法已不能完整描述观测到的染色质状态,其更接近于一个连续体,过去所说的“常染色质”和“异染色质”位于连续体两端,中间存在大量过渡。
空口无凭,这里引用Nucleus的一篇文献(Nucleus. 2022; 13(1): 236–276)中的表述:
“在细胞核内,DNA 的可及性状态多种多样,从超级可及性(通常称为“开放”染色质)到可及性较为温和的状态(称为“允许”染色质)以及更难接近或抑制的状态(称为“封闭”染色质)。开放和允许状态通常是具有转录活性的染色质,常与术语“常染色质”互换使用,而封闭状态通常称为“异染色质”。但值得注意的是,这些术语最初是用来定性描述大型基因组区域,随着对染色质复杂性的认识不断更新,常染色质和异染色质不一定是可及性的最佳描述。评估 DNA 和蛋白质之间的关系为定义染色质可及性提供了一个更有用的定量框架。构建这种关系的三种可能方式是通过:
(1)蛋白质扩散到基因组位点附近;
(2)蛋白质以非序列特异性的方式结合和/或修饰 DNA 的能力;
(3)蛋白质在特定基序上结合/修饰 DNA 的能力。”[1]
正文
根据(Nucleus. 2022; 13(1): 236–276),主要有以下技术可以应用于染色质可及性检测:[1]
MPE-seq
基于自由基的接头 DNA 切割方法,以最小的序列偏差绘制核小体和亚核小体保护的 DNA 片段。
FAIRE-seq
非核酸酶全基因组可及性测定,使用甲醛固定和超声处理来富集超可及区域。
DNase-seq
绘制基因组的可访问区域,并偏向于超可访问区域,例如增强子和启动子。
MNase-seq
可以根据酶剂量绘制超易接近区域或核小体位置。 MNase 具有基于碱基对组成的强烈切割偏差。
ATAC-seq
使用 Tn5 转座酶绘制超易接近区域图谱,该转座酶只需一步即可将测序接头切割并插入到细胞 DNA 中。可用于单细胞。
NA-seq
通过限制性内切酶和测序测量 DNA 可及性。可以在同一检测中探测基因组的超易接近区域和其他区域。分辨率取决于基因组中限制性位点的数量。
Fiber-seq
使用 N6-腺嘌呤甲基转移酶(Btr192IV、EcoGI、EcoGII、Hia5 或 Hin1523)对细胞核进行染色质足迹分析,然后进行长读长测序。
dCas9 实时跟踪
通过基于显微镜的蛋白质追踪和针对特定基因座的单分子 dCas9 颗粒的结合动力学来测量可及性。
RICC-seq
使用电离辐射和测序来探测短程染色质接触 (>1kb) 。
虽然这里写的如此繁杂,但我们接下来真正会说的只有其中四种(能考到的也只有这四种):
DNase-Seq
原理:利用类似于足迹法的方式,用DNaseI切割超敏位点,之后用新一代测序技术测序,得到被切割的超敏位点,进而反推出何者为开放染色质。
特点:①切割位点具有一定选择性:和FAIRE-Seq相比,DNase-Seq更倾向于切割调控序列区,如启动子,因此两者均存在一定程度的偏差;[2]
②消化程度高:相较于MNase-Seq,DNase可以消化DNA至10bp左右的大小。[3]
相比之下,DNase-Seq的效果在这四个里面不算太差,并且也在向单细胞方向改进,在灵敏度方面较FAIRE-Seq更高。
MNase-Seq
原理:使用核酸内切酶和微球菌核酸酶来结合和切割真核染色质DNA的蛋白质非结合区域,首先切割并切除一条链,然后也切割反平行链。染色质可以与甲醛交联或否。如果DNA区域与核小体核心(即组蛋白)或其他染色质结合蛋白(例如转录因子)结合,则MNase无法结合并切割该区域。
特点:①MNase需要Ca2+作为辅助因子,通常终浓度为1mM;
②短读长、高通量测序是MNase-seq的理想选择,因为这些技术的读长非常准确,但长度只能覆盖几百个连续碱基对;
③和DNase-Seq相比,对于DNA的切割能力不强,大概切到140bp左右为止,因此在一开始还用来作为验证核小体结构的工具之一;
④识别的特异性低,基本不会对调控序列有特殊的偏好性。[3]
总的来说,MNase-Seq的功能还是可以的,应用方向上会和Dnase等有所差异,毕竟其余三个的偏好性更强。
FAIRE-Seq
原理:该方法基于以下事实:甲醛交联在核小体结合的DNA中比在基因组的核小体耗尽区域中更有效。然后,该方法分离通常在开放染色质中发现的非交联DNA,然后对其进行测序。该方案包括交联、苯酚提取和水相DNA测序。超声处理后,使用苯酚-氯仿萃取分离片段化和固定的 DNA。该方法产生两相:有机相和水相。由于其生化特性,与核小体交联的DNA片段将优先位于有机相中。另一方面,核小体耗尽或“开放”区域将出现在水相中。通过专门提取水相,只有核小体耗尽的区域才会被纯化和富集。[4]
特点:①偏好性:据说FAIRE-seq在增强子区域的覆盖率高于启动子区域,这与DNase-seq形成对比,众所周知,DNase-seq对启动子区域表现出更高的敏感性。此外,FAIRE-seq据称显示出对内部内含子和外显子的偏好。[4]
②准确度:一般来说,人们还认为FAIRE-seq数据显示出更高的背景水平,使其成为一种不太敏感的方法。FAIRE-Seq方法依赖于甲醛交联进行观测与分离提取,交联可能会结合其他与DNA瞬时相互作用的染色质结合蛋白,进而限制了可从水相中回收和分析的非交联DNA的量。因此,从FAIRE-seq获得的整体分辨率可能相对低于DNase-seq或MNase-seq。[3][4]
③样本量要求:FAIRE-seq最少需要十万个细胞的输入。因此,MNase-Seq和ATAC-Seq等单细胞等效物使它们成为更具吸引力的替代品。[4]
FAIRE-Seq的固有优势为直接检测到开放DNA片段而无需和前面两者一样进行反推,但是分辨率低和样本量大这两个问题限制了其进一步发展应用。
ATAC-Seq
原理:通过使用高度活跃的突变体Tn5转座酶探测开放染色质,该突变体Tn5转座酶将测序接头插入基因组的开放区域。虽然天然存在的转座酶活性较低,但ATAC-seq使用突变的高活性转座酶。在一个称为“标签化”的过程中,Tn5转座酶使用测序接头切割并标记双链 DNA,然后对标记的DNA片段进行纯化、PCR扩增,并使用下一代测序进行测序。
特点:①快速高效:不需要像FAIRE-seq那样进行超声处理或苯酚-氯仿提取,没有像MNase-seq或DNase-seq这样敏感的酶消化,整个工作可在三小时内完成;
②分辨率高:相比于其他三种方法,其操作位点更为精细,因此分辨率可以达到单核苷酸水平;
abcdefg
- ↑ 1.0 1.1 [1]Andrés R. Mansisidor,Viviana I. Risca.Chromatin accessibility: methods, mechanisms, and biological insights.Nucleus.Nov.2022,13(1):238-278.doi:10.1080/19491034.2022.2143106
- ↑ [2]DNase-Seq--Wikipedia,the free encyclopedia
- ↑ 3.0 3.1 3.2 [3]MNase-seq--Wikipedia,the free encyclopedia
- ↑ 4.0 4.1 4.2 4.3 [4]FAIRE-Seq--Wikipedia,the free encyclopedia
