生物信息数据库及工具简介整理:修订间差异
Shiningstars(留言 | 贡献) |
Shiningstars(留言 | 贡献) |
||
| 第197行: | 第197行: | ||
**EMBL-EBI是指欧洲生物信息学研究所(European Bioinformatics Institute),EMBL的一部分。1982年建立了先进的核苷酸序列数据库(EMBL-DNA),可进行核苷酸序列检索及序列相似性查询。TrEMBL | **EMBL-EBI是指欧洲生物信息学研究所(European Bioinformatics Institute),EMBL的一部分。1982年建立了先进的核苷酸序列数据库(EMBL-DNA),可进行核苷酸序列检索及序列相似性查询。TrEMBL | ||
**SIB,瑞士生物信息学研究所,维护着ExPASy的服务器,Swissprot | **SIB,瑞士生物信息学研究所,维护着ExPASy的服务器,Swissprot | ||
*** '''ExPASy''' | *** '''ExPASy'''是一个集成了多种生物信息学工具和数据库的平台,专门用于蛋白质和蛋白质组学的研究。除了蛋白质相关的数据外,ExPASy还提供了其他生物信息学资源,如代谢通路数据库(KEGG京都代谢通路基因什么什么库)等。其中,Protparam工具是经常使用的功能,它可以根据蛋白质序列估计蛋白质的等电点(pi)、疏水性等特性。 | ||
**** Protscale判断蛋白质序列的疏水性,返回的结果是疏水性曲线,亲水用负值表示,疏水用正值表示。原理是滑动窗口 | |||
**** Prosite数据库是一个用于存储和提供蛋白质序列和结构特征的资源。 主要功能是提供蛋白质的功能和结构信息可以识别功能域和模式,可用于蛋白质家族和亲缘关系的研究,预测蛋白质结构与功能 | |||
** PIR,由美国国家生物医学研究基金会(NBRF)于1984年成立,用于支持基因组学、蛋白质组学和系统生物学研究和科学研究。提供蛋白质数据库和分析工具,包括蛋白质序列数据库 (PSD)。PIR-PSD | ** PIR,由美国国家生物医学研究基金会(NBRF)于1984年成立,用于支持基因组学、蛋白质组学和系统生物学研究和科学研究。提供蛋白质数据库和分析工具,包括蛋白质序列数据库 (PSD)。PIR-PSD | ||
*数据库的关系 | *数据库的关系 | ||
| 第211行: | 第213行: | ||
[https://zhuanlan.zhihu.com/p/599641613 感谢知乎qwq] | [https://zhuanlan.zhihu.com/p/599641613 感谢知乎qwq] | ||
*Uniprot内部有UniPrac,Uniref,UniProtKB三层,由粗糙到精细列 | |||
** 第一层次:UniPrac(Uniprot archive,档案馆):三个子库中所有序列的直接合并,信息比较粗糙,冗余。UniParc由于数据冗余,将相同序列合并一条,唯一编号。 | ** 第一层次:UniPrac(Uniprot archive,档案馆):三个子库中所有序列的直接合并,信息比较粗糙,冗余。UniParc由于数据冗余,将相同序列合并一条,唯一编号。 | ||
** 第二层次:UniRef(UniProt Reference Clusters):去除了重复序列,UniRef100即去除了完全相同的冗余序列后的剩余,UniRef90即去除了相似性在90%以上的相似序列后的剩余,依此类推。 | ** 第二层次:UniRef(UniProt Reference Clusters):去除了重复序列,UniRef100即去除了完全相同的冗余序列后的剩余,UniRef90即去除了相似性在90%以上的相似序列后的剩余,依此类推。 | ||
** 第三层次:UniProtKB(UniProt KnowledgeBase):详细注释的,有文献和其他数据库链接的精品数据库,有UniProtKB/TrEMBL(自动注释)和UniProtKB/Swissprot(人工注释)两部分。 | ** 第三层次:UniProtKB(UniProt KnowledgeBase):详细注释的,有文献和其他数据库链接的精品数据库,有UniProtKB/TrEMBL(自动注释)和UniProtKB/Swissprot(人工注释)两部分。<TrEMBL和Swissprot热血沸腾的组合技> | ||
* 还有Proteomes库是蛋白质组数据库 | * 还有Proteomes库是蛋白质组数据库 | ||
| 第261行: | 第264行: | ||
* SCOPe引入自动化分类,但核心仍保留手动注释。 | * SCOPe引入自动化分类,但核心仍保留手动注释。 | ||
<这段deepseek写的,我对着笔记查了一下> | |||
<references /> | <references /> | ||
2025年3月16日 (日) 22:16的版本
现介绍生物信息数据库及工具。
国际核苷酸序列数据库协作 (INSDC),包含NLM-NCBI(美国),ENA(欧洲,EMBL与EBI参与),DDBJ(日本),
其创始成员是信息和系统研究组织、日本国家遗传学研究所 (ROIS-NIG)、欧洲分子生物学实验室 - 欧洲生物信息学研究所 (EMBL-EBI) 和美国国立卫生研究院的一个组成部分,国家医学图书馆-国家生物技术信息中心 (NLM-NCBI)。
NCBI(National Center for Biotechnology Information,美国国家生物技术信息中心)
已故的参议员克劳德·佩珀(Claude Pepper)认识到计算机信息处理方法对于进行生物医学研究的重要性,并发起了立法。他于1988年11月4日在美国国立卫生研究院(NIH)成立了美国国家生物技术信息中心(NCBI)并作为美国国家医学图书馆(NLM)的一个部门。选择了在NLM是因为它有创建和维护生物医学数据库方面的经验,又因为它是NIH的一部分,因此可以建立计算分子生物学的研究计划。 NIH的集体研究组成了世界上最大的生物医学研究机构。(机翻)
(原文:The late Senator Claude Pepper recognized the importance of computerized information processing methods for the conduct of biomedical research and sponsored legislation that established the National Center for Biotechnology Information (NCBI) on November 4, 1988, as a division of the National Library of Medicine (NLM) at the National Institutes of Health (NIH). NLM was chosen for its experience in creating and maintaining biomedical databases, and because as part of NIH, it could establish an intramural research program in computational molecular biology. The collective research components of NIH make up the largest biomedical research facility in the world.)[1]
PubMed诞生于1996年。最常用的文摘型数据库。其收录范围不仅包括Medline,还有大部分PMC和NIH基金资助作者的文献,NCBI书籍等。PubMed还提供链接到文献全文所在网站的全文链接。早期 PubMed 数据库主要的任务,是将 Mesh 目录(MEDlINE的早期目录,主题词表)中收录的医学主题与大量有关医学研究的文献相匹配。这意味着,PubMed最早的发布时间应该是在 1971 年,也就是 MEDLINE 数据库第一次正式发布的时间。
MEDLINE始建于1966年(追溯收录至1946年),是NLM的期刊文献记录数据库。1983年,MEDLINE推出电子版,网上数据每日更新,光盘版每月更新。MEDLINE有严格的文献选择委员会LSTRC进行选刊。目前MEDLINE的文献都收录在PubMed中。
PMC全称为PubMed Central,建立于2000年,由NCBI进行管理,是可以免费获取全文的生物医学和生命科学全文数据库,保存了NLM收录印刷杂志的电子副本。PMC的部分bookreview内容没有收录于PubMed。
Entrez是NCBI开发的检索系统,可以查Genbank,PubMed(文献数据库)等数据库的信息。
Genbank是由NCBI建立的DNA数据库,包含EST(表达序列标签)和STS(序列标记位点)等。
- GBFF是Genbank的原始文件格式,包含很多注释,
- LOCUS表示基因座名字,核酸序列长度,分子类别,拓扑类型,更新日期
- ACCESSION检索号,数据在数据库当中唯一不变的检索编号,用GCA_起始
- VERSIONS版本号格式为“检索号.数字”,初始为1,更改时版本号增加(从2015开始NCBI淘汰了写在版本号旁边的改一次变一次的GI
- FEATs(FEATUREs)重要注释内容<其中包含dna来源质粒/基因组?的source,RBS,CDS,promoter启动子)
- SOURCE表示来源生物物种的俗名,如小鼠和人
- KEYWORDS用于检索的关键词
- REFERENCE参考文献
- ORIGRINS核酸序列 ATCTTGCATAC,用“//”双斜线结束
- FASTA使用于序列比对的,格式第一部分是序列名字,与>相连,第二部分用空格与序列名字相隔,表示注释信息,可以没有。第三部分bp一行60字母
- FASTQ测序仪原始数据,共四行
- 第一行: 用“@”开头,写read的名称
- 第二行: 测序序列,ATCGN(识别不出来的时候写N
- 第三行:用“+”开头,描述
- 第四行: 用ASCII码表示的read质量分数 数据库
- Refseq,来自NCBI的参考序列计划,提供非冗余的标准序列,包括基因组,蛋白质和染色体
- dBEST,表达序列标签库
BLAST是由ncbi开发的序列比对工具,基于局部序列比对,寻找序列之间的相似者
- 返回值
- E值Expect,∈[0,1],表示随机配对的可能性,E值越大,随机可能性越大
- 相似性/一致性Identities,匹配上的碱基数占总序列长的百分数。
- score得分,来自打分矩阵,分数高同源性强
- Gaps两序列之间缺少或多出的对不上部分,缺失或插入,用——表示
- Strand
- plus/plus +/+表示两条序列是相同方向的
- plus/minus +/-表示两条序列一正一反,5-3与3-5
- 类型
- BLASTn,对比核酸及其互补序列与核酸库
- BLASTp,对比蛋白质与蛋白库
- BLASTx,将核酸翻译为蛋白质后比对
- tBLASTn,将蛋白质翻译为核酸后比对核酸
- tBLASTx,将核酸翻译成蛋白质后与数据库中翻译成蛋白质的核酸进行比对
数据库
Assembly数据库
标签:综合基因组数据库
一个提供有关组装的基因组结构,组装名称和其他元数据,统计报告以及与基因组序列数据的链接的信息的数据库。(机翻)
(原文:A database providing information on the structure of assembled genomes, assembly names and other meta-data, statistical reports, and links to genomic sequence data.)
Assembly数据库包含有关组装基因组结构的信息,如AGP文件中所示或完整测序的染色体的集合。 该数据库提供了版本化的程序集登录号,该编号可跟踪程序集的更改,这些更改是通过随着时间的推移提交组来更新的。 该Web资源提供有关程序集的元数据,例如程序集名称(和备用名称),简单的程序集统计报告(重叠群,脚手架的类型和数量; N50)以及更新的历史视图。 它还跟踪提交给国际核苷酸序列数据库协作(INSDC)的程序集(即DDBJ,ENA或GenBank)与NCBI参考序列(RefSeq)项目中表示的程序集之间的关系。(机翻)
(原文:The Assembly database has information about the structure of assembled genomes as represented in an AGP file or as a collection of completely sequenced chromosomes. The database provides a versioned Assembly accession number that tracks changes to assemblies as they are updated by submitting groups over time. The web resource provides meta-data about assemblies such as assembly names (and alternate names), simple statistical reports of the assembly (type and number of contigs, scaffolds; N50s) and a history view of updates. It also tracks the relationship between an assembly submitted to the International Nucleotide Sequence Database Collaboration ( INSDC ), i.e. DDBJ , ENA or GenBank , and the assembly represented in the NCBI Reference Sequence (RefSeq) project.)
网页视图:
网页地址:https://www.ncbi.nlm.nih.gov/assembly[2][3]
BioCollections数据库
标签:标本数据库
是用于文化收藏,博物馆,草本植物和其他自然历史收藏(包括达尔文核心机构和收藏代码)的链接元数据的精选数据集,以及用于将标本ID映射到收藏站点上的网页的链接。Biocollections存储“结构化凭证”(机构代码:可选集合代码:样本ID,如:/culture_collection="ISBC:CMF:1866")中使用的首字母缩略词,用于向国际核苷酸序列数据库(INSDC)(GenBank,欧洲核苷酸档案库(ENA)和日本DNA数据库(DDBJ))和NCBI的BioSample提交的序列条目。(机翻)
(原文:BioCollections is a curated dataset of metadata for culture collections, museums, herbaria and other natural history collections, including Darwin Core institution and collection codes, and URL formulae for mapping specimen ids to web pages at the collection site. Biocollections stores acronyms used in “structured vouchers” for sequence entries submitted to the International Nucleotide Sequence Database (INSDC)(GenBank, European Nucleotide Archive (ENA), and DNA Databank of Japan (DDBJ)) and NCBI’s BioSample.)
注意:本数据库不收录来自个人的生物标本藏品,仅指向各馆藏数据库。
查询:
| 代码 | 意思 | 注文 | |
|---|---|---|---|
| [icode] | 检索机构代码(部分代码) | ||
| [uicode] | 检索唯一的机构代码 | ||
| [ccode] | 检索对应的收藏代码(/specimen_voucher="UAM:Mamm:24119") | 哺乳类:Mamm
鱼类:Fish 昆虫:Ento | |
| [iname] | 搜索机构名称 | ||
| [cname] | 搜索收藏类型 | ||
| [all] | 以上所有 | ||
| 按类别搜索 | collection type museum[prop] | 检索博物馆 | |
| collection type herbarium[prop] | 检索植物标本室 | ||
| collection type culture collection[prop] | 检索文化类型收集 | ||
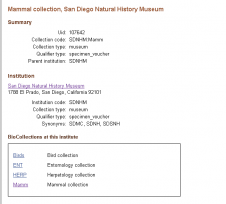
网页视图:
网页地址:https://www.ncbi.nlm.nih.gov/biocollections[2][4][5]
BioProject数据库(旧称:Genome Project)
标签:项目计划数据库
生物项目是与来自单个组织或财团的单个计划相关的生物数据的集合。 BioProject记录为用户提供了一个单一的位置,可以找到该项目生成的各种数据的链接,并存储到INSDC成员维护的档案数据库中。 BioProject的典型示例包括用于对多种细菌菌株进行测序的多分离项目,或特定生物的基因组和转录组的单分离项目。您提供的有关这项研究工作的描述对于为实验数据提供上下文非常重要。一个基因组学,功能基因组学和遗传学研究的集合,并链接到它们产生的数据集。该资源描述了项目的范围,材料和目标,并提供了一种检索数据集的机制,这些数据集由于注释不一致,多次独立提交以及通常存储在不同数据库中的多种数据类型的不同性质而常常很难找到。(机翻)
(原文:A BioProject is a collection of biological data related to a single initiative originating from a single organization or from a consortium. A BioProject record provides users a single place to find links to the diverse data generated for that project and deposited into the archival databases maintained by members of the INSDC. Typical examples of a BioProject include a multiisolate project for sequencing multiple strains of a bacterial species, or a monoisolate project for the genome and transcriptome of a particular organism. The description you supply about this research effort is important for providing context to your experimental data. A collection of genomics, functional genomics, and genetics studies and links to their resulting datasets. This resource describes project scope, material, and objectives and provides a mechanism to retrieve datasets that are often difficult to find due to inconsistent annotation, multiple independent submissions, and the varied nature of diverse data types which are often stored in different databases.)
大型计划:
The 1000 Genomes Project (human)千人基因组计划(ID:28889)
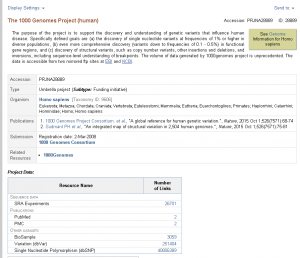
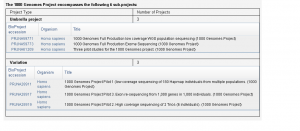
The human ENCODE (ENCyclopedia Of DNA Elements) project人类DNA元素百科全书计划(ID:30707)
NIH Human Microbiome Project (HMP) Roadmap Project人类微生物组计划(ID:43021)
浏览计划样式:
网页视图:
网页地址:https://www.ncbi.nlm.nih.gov/bioproject[2]
BioSample数据库
标签:实验材料数据库
BioSystems数据库
UCSC基因组浏览器
UCSC 基因组浏览器是由加州大学圣克鲁斯分校(UCSC)的在线可下载基因组浏览器。提供来自各种脊椎动物和无脊椎动物物种以及主要模式生物的基因组序列数据,是一个图形查看器.
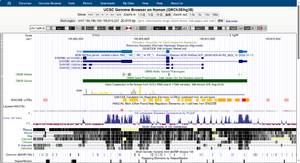
可以看到,UCSC浏览器上可以看到各个数据库对同一基因组片段的注释,而这里恰好是音猬因子SHH所在处。自上而下有:
- 蓝色的GENCODE
- 深蓝色的RefSeq
- 绿色的OMIM(在线人类孟德尔遗传),收集了有关人类遗传病的突变。
- 黄色的ENCODE
- 等等等等

关于RefSeq的条带,四条条带代表四个剪接变体,细线是被剪掉的内含子,较细的粗线是UTR,较粗的粗线是蛋白质编码序列。细线上的小箭头是转录方向的意思。
BLAT
BLAT(BLAST-like比对工具)也出自UCSC,最初目的是快速的将小鼠基因组和人类基因组对齐,速度提升了数百倍。
EMBL(European Molecular Biology Laboratory)欧洲分子生物学实验室
总部位于德国海德堡,1974年由欧洲14个国家加上亚洲的以色列共同发起建立,现在由欧洲29个成员国政府支持组成,目的在于促进欧洲国家之间的合作来发展分子生物学的基础研究和改进仪器设备、教育工作等。
UniProt:联合蛋白质序列数据库
- 起源:TrEMBL、Swiss-Prot、PIR-PSD三个数据库的数据合并而成.2002 年,PIR 与其国际合作伙伴 EBI(欧洲生物信息学研究所)和 SIB(瑞士生物信息学研究所)一起获得了 NIH 的资助,创建了 UniProt。 通过统一 PIR-PSD、Swiss-Prot 和 TrEMBL 数据库,实现蛋白质序列和功能的单一全球数据库。
- Uniprot提供亚细胞定位,翻译后修饰
- Uniprot是包含蛋白质序列,功能信息,论文索引的蛋白质数据库,整合了包括EBI,SIB,PIR三大数据库的资源。
- EMBL-EBI是指欧洲生物信息学研究所(European Bioinformatics Institute),EMBL的一部分。1982年建立了先进的核苷酸序列数据库(EMBL-DNA),可进行核苷酸序列检索及序列相似性查询。TrEMBL
- SIB,瑞士生物信息学研究所,维护着ExPASy的服务器,Swissprot
- ExPASy是一个集成了多种生物信息学工具和数据库的平台,专门用于蛋白质和蛋白质组学的研究。除了蛋白质相关的数据外,ExPASy还提供了其他生物信息学资源,如代谢通路数据库(KEGG京都代谢通路基因什么什么库)等。其中,Protparam工具是经常使用的功能,它可以根据蛋白质序列估计蛋白质的等电点(pi)、疏水性等特性。
- Protscale判断蛋白质序列的疏水性,返回的结果是疏水性曲线,亲水用负值表示,疏水用正值表示。原理是滑动窗口
- Prosite数据库是一个用于存储和提供蛋白质序列和结构特征的资源。 主要功能是提供蛋白质的功能和结构信息可以识别功能域和模式,可用于蛋白质家族和亲缘关系的研究,预测蛋白质结构与功能
- ExPASy是一个集成了多种生物信息学工具和数据库的平台,专门用于蛋白质和蛋白质组学的研究。除了蛋白质相关的数据外,ExPASy还提供了其他生物信息学资源,如代谢通路数据库(KEGG京都代谢通路基因什么什么库)等。其中,Protparam工具是经常使用的功能,它可以根据蛋白质序列估计蛋白质的等电点(pi)、疏水性等特性。
- PIR,由美国国家生物医学研究基金会(NBRF)于1984年成立,用于支持基因组学、蛋白质组学和系统生物学研究和科学研究。提供蛋白质数据库和分析工具,包括蛋白质序列数据库 (PSD)。PIR-PSD
- 数据库的关系
通过INSDC等得到原始数据,处理后存入UniParc。
UniParc作为数据仓库,再分别给UniProtKB,Proteomes,UniRef提供可靠的数据集。 这里的UniProtKB 由两个子库构成 Swiss-Prot,TrEMBL。
Swiss-Prot 经过人工验证和注释,是高质量的蛋白质注释数据。但人工效率在高速增长的蛋白质数据面前显得极其低效。因此,注释这些数据需要大量时间,为了弥补这一问题。TrEMBL 被建立用于存储由机器自动翻译和预测的蛋白质序列。导致蛋白质数据冗余↑可信度↓。
简而言之,在UniProtKB数据库中Swiss-Prot是由TrEMBL经过手动注释后得到的高质量非冗余数据库,也是我们今后常用的蛋白质数据库之一。
- Uniprot内部有UniPrac,Uniref,UniProtKB三层,由粗糙到精细列
- 第一层次:UniPrac(Uniprot archive,档案馆):三个子库中所有序列的直接合并,信息比较粗糙,冗余。UniParc由于数据冗余,将相同序列合并一条,唯一编号。
- 第二层次:UniRef(UniProt Reference Clusters):去除了重复序列,UniRef100即去除了完全相同的冗余序列后的剩余,UniRef90即去除了相似性在90%以上的相似序列后的剩余,依此类推。
- 第三层次:UniProtKB(UniProt KnowledgeBase):详细注释的,有文献和其他数据库链接的精品数据库,有UniProtKB/TrEMBL(自动注释)和UniProtKB/Swissprot(人工注释)两部分。<TrEMBL和Swissprot热血沸腾的组合技>
- 还有Proteomes库是蛋白质组数据库
蛋白质结构数据库
一级结构数据库
PDB蛋白质结构数据库(Protein Data Bank)是美国Brookhaven国家实验室于1971年创建的,由结构生物信息学研究合作组织(RCSB)维护。
- PDB数据库以文本文件形式存储分子数据,每个分子形用一个独立的文件。早期的分子文件的文件名后缀为“.pdb”,1997年以后每1种生物大分子对应1组(3个)相关文件分别是:全文文件(后缀为“.full”相当于原来的“.pdb”文件)、书目文件(后缀为“.biblio”)和图形文件(后缀为“.gif”)。
- PDB数据库是生物大分子的原子坐标和描述蛋白质和其他重要生物大分子相关信息的存储库。结构生物学家使用x 射线晶体学、核磁共振光谱和冷冻电子显微镜等方法来确定分子中每个原子相对于彼此的位置,然后存储这些信息,并由PDB数据库注释并公开发布到档案中。
二级结构数据库
DSSP工具:DSSP程序是一个二级结构分配(以及更多信息)的数据库,涵盖PDB中的所有蛋白质条目。DSSP也是从PDB条目计算DSSP条目的程序。DSSP并不预测二级结构。
- 根据氢键模式和主链构象,将蛋白质的每个残基分类为特定的二级结构类型。算残基之间的氢键能量,用于确定二级结构的边界。计算每个残基的溶剂可及表面积(ASA),反映其在蛋白质表面的暴露程度。
- 输入:蛋白质的三维结构文件(通常为PDB格式)。输出:包含每个残基的二级结构类型、氢键信息、溶剂可及性等数据的文本文件。
Pfam数据库重视蛋白质结构域和家族(基于序列同源性),定期通过算法扩展新家族。
- 关注功能相关的蛋白质区域(如结构域),而非完整蛋白质的结构。
- 分类依据:
- 序列相似性,通过隐马尔可夫模型(HMM)比对识别保守区域。
- 基于多序列比对和进化关系。
- 层次结构:
- 超家族(Clan):多个家族在进化或功能上的关联。
- 家族(Family):具有显著序列相似性的结构域集合。
CATH数据库提供蛋白质三维结构与分类,关注结构的拓扑和进化起源。
- 蛋白质三维结构的层次化分类(从二级结构到进化关系)以三维结构特征为依据,结合序列和功能信息进行分类
- 分类层次:
- Class(类别):基于二级结构组成(如全α、全β、α+β、α/β)。
- Architecture(架构):描述二级结构的空间排布(如β桶、α螺旋束)。
- Topology(拓扑):基于折叠方式和连接性。
- Homologous Superfamily(同源超家族):进化相关的蛋白质。
SCOP数据库分类强调进化同源性,基于结构与进化关系分类,由专家手动分类,强调超家族内的远缘同源关系。
- 分类层次:
- Class(类别):基于二级结构类型(如全α、全β等)。
- Fold(折叠方式):具有相似拓扑结构的蛋白质。
- Superfamily(超家族):推测具有共同祖先的结构相似蛋白质。
- Family(家族):明确序列同源性的蛋白质集合。
- SCOPe引入自动化分类,但核心仍保留手动注释。
<这段deepseek写的,我对着笔记查了一下>
