基因组结构变异的检测方法:修订间差异
外观
创建页面,内容为“1” |
无编辑摘要 |
||
| 第1行: | 第1行: | ||
1 | [[文件:基因组结构变异的检测方法.webp|无框]] | ||
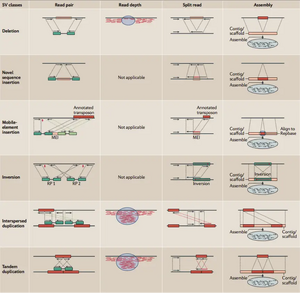
=== 1.RP(read pair) === | |||
==== 原理: ==== | |||
利用双端测序(Paired-End Sequencing)数据中读段对的空间关系异常来推断大片段变异。 | |||
一般来说我们是无法直接获得每一对read1和read2之间真实的插入片段长度的,但通过序列比对,计算它们彼此之间比对位置上的距离却可以间接获得这个长度。正常情况下这个长度应该等于我们测序时的插入片段长度的,如果出现了数据偏离,就可以判断在read上出现了变异(线性变异,特指deletion和insertion) | |||
而通过比对read1和read2之间的序列位置关系,还能够发现更多非线性的序列变异。比如,序列倒置(Inversion),因为,按照PE的测序原理(其实就是中间有插入片段版本的illumina测序),read1和read2与参考基因组相比对,正好是一正一负,要么是read1比上正链,read2比上负链,要么是反过来,而且read1和read2都应处于同一个染色体上,如果不是这种现象,那么就很可能是序列的非线性结构性变异所致(倒位和易位) | |||
==== 缺陷: ==== | |||
# 检测deletion时,如果变异的碱基对数量较小,难以检测出来(因为我们用RP检测的时候会要求插入片段长度的变化具有统计意义上的显著性,所以它所能检测到的片段长度就会受插入片段长度的标准差SD所影响) | |||
# 所能检测的insertion长度不能超过插入片段,因为我们去计算插入片段长度时是要把得到的read1-插入片段-read2放回基因组里去比对的,但如果insertion片段太长了,那么read根本不会匹配上基因组,也就是说,你根本不知道这里会有一段序列 | |||
2025年5月30日 (五) 22:13的版本
1.RP(read pair)
原理:
利用双端测序(Paired-End Sequencing)数据中读段对的空间关系异常来推断大片段变异。
一般来说我们是无法直接获得每一对read1和read2之间真实的插入片段长度的,但通过序列比对,计算它们彼此之间比对位置上的距离却可以间接获得这个长度。正常情况下这个长度应该等于我们测序时的插入片段长度的,如果出现了数据偏离,就可以判断在read上出现了变异(线性变异,特指deletion和insertion)
而通过比对read1和read2之间的序列位置关系,还能够发现更多非线性的序列变异。比如,序列倒置(Inversion),因为,按照PE的测序原理(其实就是中间有插入片段版本的illumina测序),read1和read2与参考基因组相比对,正好是一正一负,要么是read1比上正链,read2比上负链,要么是反过来,而且read1和read2都应处于同一个染色体上,如果不是这种现象,那么就很可能是序列的非线性结构性变异所致(倒位和易位)
缺陷:
- 检测deletion时,如果变异的碱基对数量较小,难以检测出来(因为我们用RP检测的时候会要求插入片段长度的变化具有统计意义上的显著性,所以它所能检测到的片段长度就会受插入片段长度的标准差SD所影响)
- 所能检测的insertion长度不能超过插入片段,因为我们去计算插入片段长度时是要把得到的read1-插入片段-read2放回基因组里去比对的,但如果insertion片段太长了,那么read根本不会匹配上基因组,也就是说,你根本不知道这里会有一段序列