各种工具酶:修订间差异
无编辑摘要 |
无编辑摘要 |
||
| 第68行: | 第68行: | ||
=== 限制性内切酶的星号活性 === | === 限制性内切酶的星号活性 === | ||
星号活性指一定条件下限制性酶切开了序列类似但不同的位点。原因可能是: | |||
* | * 甘油浓度过高。限制性内切酶都是在50%的甘油中保存的,最终反应体系甘油浓度不应超过5%(即酶不应超过10%)。 | ||
* 酶浓度过高。 | |||
* 离子强度过低。 | |||
* pH过高。 | |||
* 有机溶剂存在。 | |||
* 不合适的二价阳离子。 | |||
== DNA连接酶 == | == DNA连接酶 == | ||
2024年8月1日 (四) 23:52的版本
限制性内切酶
限制性内切酶在各种细菌和古菌中都具有(大家都怕噬菌体);限制-修饰系统将自身基因组中特定序列甲基化,然后切割没有甲基化的“入侵者”.
限制性内切酶的种类
Ⅰ型限制性内切酶
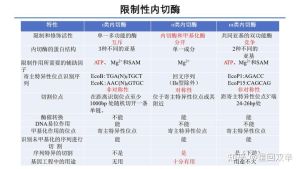
功能:切割无甲基化的外来DNA;修饰半甲基化的DNA(刚刚完成复制的基因组DNA)为全甲基化的DNA。
识别位点:有两段识别位点,中间隔着一段非特异序列。比如识别TGANNNNNNNNTGCT的EcoB。不是回文序列。
切割位点:这类酶结合位点后会在DNA链上开始移动,直到数kb外的随机位点切割。显然,他们对科研没什么用处。
组成:三个亚基,分别负责切割、甲基化等功能。
辅因子:SAM、ATP、Mg
Ⅱ型限制性内切酶
功能:只能切割,不能甲基化。
识别位点:回文序列(除了ⅡS类),多数是6bp,也有4和8的,甚至还有5(EcoRⅡ)和7(BbvCⅠ)的。
切割位点:在回文序列内部(除了ⅡS类),酶不会在DNA上迁移。最有用的一类
组成:同源二聚体
辅因子:仅Mg
Ⅲ型限制性内切酶
功能:切割和甲基化
识别:5~6bp的非回文序列
切割位点:序列旁边25~27bp的特定位点(就是说距识别位点距离是固定的,不像Ⅰ)。也没有什么用。
辅因子:SAM,ATP,Mg
ⅡS型限制性内切酶
识别非回文序列,切割位点紧邻非回文序列,可用于GoldenGate克隆。
限制性内切酶的命名
举个例子:EcoRⅠ
E,代表E.coli的属名,单字母,大写,斜体。
co,代表E.coli的种加词,两字母,小写,斜体。
R,代表来自大肠杆菌菌株R,一字母,正体,有时大写有时小写——如HindⅢ的d代表来自Rd菌株。
Ⅰ,代表是在该菌株中发现的第一种限制性内切酶。
同尾酶和同裂酶
同尾酶:切割后露出的粘性末端一致,但识别位点不相同的酶。两种酶切除的末端链接后,谁也不能再次切开。
同裂酶:识别序列相同,但切割位点和露出粘性末端的序列未必相同的酶
如何计算限制性内切酶的切割片段平均长度
如果识别一个六bp回文序列,那么平均4^6bp会有一个位点。
如果识别的序列中仅限制是嘌呤或者是嘧啶,比如识别ARGCYT,那就是4*2*4*4*2*4bp有一个。
- 好像很简单,但这里其实要绕一个弯子:在一条链上出现“AAATTT”的概率是每4^6bp一次,那另一条链上出现的概率也是4^6bp一次,加起来应该每4^6÷2bp出现一次啊?这是因为我们恰好计算了一个回文序列,如果一个位点是AAATTT,那么他的对面也应该是AAATTT.反过来,一个位点不是AAATTT,他的对面一定不是AAATTT。因此我们不用计算两条链。但如果我们计算了一个非回文序列,那么就要除以二了。以上是个人思考,没有在题目中见过,也没有佐证的材料,望谨慎看待。
几种限制性内切酶的介绍
- NotⅠ,识别GCGGCCGC,因此切割位点特别稀少,称为稀切点酶。有用处。
- dpnⅠ:识别GA/TC, 只有当A被甲基化时才能切割。可用于点突变时去除甲基化的未突变的内源质粒.
限制性内切酶的星号活性
星号活性指一定条件下限制性酶切开了序列类似但不同的位点。原因可能是:
- 甘油浓度过高。限制性内切酶都是在50%的甘油中保存的,最终反应体系甘油浓度不应超过5%(即酶不应超过10%)。
- 酶浓度过高。
- 离子强度过低。
- pH过高。
- 有机溶剂存在。
- 不合适的二价阳离子。
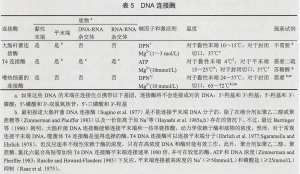
DNA连接酶
可以连接3-OH Pi-5的缺口,不能连接3-OH HO-5,也不连接3-Pi HO-5。
这个性质可用来防止载体的自环化。比如你有一条DNA序列和一条被切为线性的载体,现在要加入连接酶把二者连接。好巧不巧你使用了单酶切法,这导致载体和目标序列的粘性末端都是互补的,直接加入DNA连接酶的话,会产生很多载体自身环化的产物。他们进入细菌后,没有带来产物,却带来了抗性基因,使得能在选择培养基上生长。
为了避免这个情况,你可以先用碱性磷酸酶去除载体的磷酸,这样自环化时,就成了3-OH HO-5,不能发生;而与目标基因链接时,一条链是3-OH Pi-5,可以链接,一条链是3-OH HO-5,不能连接。这样产生有两个缺口的环状质粒,在转入大肠杆菌后,大肠杆菌会帮你连接上的。
常用的有两种:大肠杆菌连接酶和T4连接酶。关于他们的区别请看右图。
最重要的:大肠杆菌用NADH,T4噬菌体用ATP。大肠杆菌不能连接平末端,T4可以。
DNA聚合酶
DNA聚合酶Ⅰ
有三种活性:5→3外切酶。3→5外切酶。DNA聚合酶。
用途:一边切除5段,一边合成3段,令其使用放射性底物,就可以用放射性核苷酸置换普通核苷酸,产生有放射性的/被标记的探针。
Klenow片段
用枯草杆菌蛋白酶切割DNApolⅠ,产生一个小片段-有5→3切除活性和一个大片段-称为Klenow片段,具有合成酶和3→5切除活性——丧失了5→3外切酶活性不能用于探针标记。
用途:①补平3凹入-聚合活性②切除3凸出-3切活性③Sanger测序④没有发现taq酶时,PCR使用klenow片段。只不过每次加热变性后要重新加酶。
Taq酶
大名鼎鼎的taq酶,没有3外切酶活性,不能够校对。会在产物的末端额外加一个A,这个特殊的性质可用于TA克隆。
顺口一提,产生taq酶的水生栖热菌虽然生存在高温泉水中,却是一种细菌而非古菌。
Pfu酶
具有校对能力,同时还耐高温。确实来自古菌-火球菌。
反转录酶
各种核酸酶
DNaseⅠ
镁离子存在时切dsDNA一条链形成缺口,锰离子存在时切两条链成断段。
绿豆核酸酶
内切单链dna和rna,需要锌。
核酸酶s1
同样内切单链dna和rna,需要锌。
核酸酶H
内切杂交链中的RNA链。
